Для
таблиц данных, описывающих промоторы, с
помощью "Gene Discovery" проверялись
гипотезы о связи контекстных сигналов и
комплексных контекстных сигналов с
функциональным классом последовательности.
В результате работы программы для
исследованных выборок промоторов
обнаружено большое число комплексных
сигналов. Число найденных сигналов
варьировало от нескольких десятков до
тысяч, в зависимости от жесткости условий
отбора (порогового значения точного
критерия Фишера).
На
последнем шаге был проведен отбор
комплексных сигналов, удовлетворяющих
следующим дополнительным условиям:
(1) индивидуальные сигналы, входящие в
комплексный сигнал, не пересекаются на
последовательностях рассмотренных
промоторов;
(2) наблюдаемое количество промоторов N,
в которых встретился комплексный сигнал
выше числа N*, которое ожидается по
случайным причинам, N>N*.
Примеры
комплексных сигналов, удовлетворяющих этим
условиям, специфичных для промоторов генов
эндокринной системы приведены в таблице 3.4 (Витяев
и др., 2001).
Таблица
Примеры
комплексных сигналов в промоторах генов
эндокринной системы
|
№ пп |
Комплексный
сигнал1 |
Условная вероятность
сигнала в промоторах2 |
Оценка вероятности
сигнала в промоторах по критерию
Фишера3 |
Число промо-торов, имеющих
сигнал4 |
Ожидаемое по случайным
причинам количество промоторов5 |
|
1 |
CWGNRGCN<NGSYMTAM<CAGGRNCH |
0.875 |
0.00054 |
4 |
0.24 (<1) |
|
2 |
KGRSSAGR<CYCYNSCY<CWGSNYCH |
1.0 |
0.00012 |
4 |
0.28 (<1) |
|
3 |
CWGNRGCN<NGSYMTAM<MAGKSHCN |
1.0 |
0.00009 |
6 |
0.47 (<1) |
|
4 |
CWGNRGCN<NGSYMTAM<CMDGGNCH |
0.846 |
0.00099 |
5 |
0.43 (<1) |
|
5 |
CNKSAGNT<NCARGRNC<HNNKGCTG |
1.0 |
0.01426 |
4 |
0.37 (<1) |
|
6 |
RNWGGCCN<DGRGNRGG<TCMAGNMN |
0.875 |
0.00118 |
4 |
0.40 (<1) |
|
7 |
RGSNRGRG<NNGSTWTA<CNCNRKGC |
1.0 |
0.02852 |
5 |
0.53 (<1) |
|
8 |
NNGSTWTA<NMAGDGMC<CNCNRKGC |
0.875 |
0.04755 |
5 |
0.53 (<1) |
|
9 |
RGSNRGRG<NNGSTWTA<CMDGGNCH |
1.0 |
0.03964 |
5 |
0.55 (<1) |
|
10 |
RGSNRGRG<KGGNSAGD<ANCTSMNG |
1.0 |
0.03964 |
4 |
0.45 (<1) |
|
... |
... |
... |
... |
... |
... |
|
45 |
RGSNRGRG<NGSYMTAM<CNCNRKGC |
1.0 |
0.03964 |
5 |
0.58 (<1) |
Примечания. Данные в таблице приведены не полностью
из-за большого объема, пропуски обозначены
многоточиями.
1 – Комплексный
сигнал состоит из олигонуклеотидов в 15-буквенном
алфавите, линейно расположенных на
последовательности в соответствии с
приведенной записью. Знак "<"
означает, что позиция первого
олигонуклеотида относительно старта
транскрипции меньше позиции второго.
Расстояние между отдельными сигналами не
фиксировано.
2 – Условная
вероятность PC(N1,N2) считается как
отношение числа промоторов имеющих данный
сигнал N1 к общему числу
последовательностей, имеющих данный сигнал
N1/(N1+N2).
3 – Оценка вероятности получить сигнал в
промоторах по случайным причинам большее
число раз, чем наблюдаемое, по точному
критерию Фишера для таблиц сопряженности P(N1,N2,N3,N4).
4 – Количество
промоторов в обучающей выборке, имеющих
данный комплексный сигнал.
5 – Ожидаемое по случайным причинам
количество промоторов, имеющих комплексный
сигнал. В предположении независимости
входящих в комплексный сигнал
олигонуклеотидов оценивается как
произведение общего числа промоторов на
частоты олигонуклеотидов в промоторах, с
учетом вариантов их взаимного линейного
расположения.
Ожидаемое
количество N* оценивалось как
произведение частот отдельных
олигонуклеотидов в промоторах, умноженное
на общее число промоторов, с учетом числа
вариантов взаимного расположения
олигонуклеотидов на последовательности
промотора. Например, ожидаемое количество
промоторов N*, в которых встретился
комплексный сигнал (S1,S2,S3|Pos(S1)<Pos(S2)<Pos(S3)),
равно
N*=P(S1)´P(S2)
´P(S3)
´M / 6,
где N* – ожидаемое
количество промоторов, в которых
встретился комплексный сигнал; P(S1), P(S2),
P(S3) – частоты промоторов, содержащих
олигонуклеотиды S1,S2 и S3,
соответственно; M –
полное количество промоторов в
анализируемой выборке; 6=3!
– число возможных вариантов взаимного
линейного размещения трех
олигонуклеотидов в промоторе.
В
частности, для сигнала CWGNRGCN<NGSYMTAM<MAGKSHCN
в промоторах
эндокринной системы ожидаемая величина N*=0.47,
т.е. меньше единицы, в то время как он
встретился в 6 промоторах, что в
приблизительно в 13 раз больше ожидаемого
уровня (см. таблицу 3.4, третья строка). Сигнал
DNMYTTSA<DNYAADGG<RCAGMMDY в промоторах эритроидных генов имеет N*
=0.54, но встретился в 8 промоторах, что
примерно в 14 раз выше ожидаемого уровня.
Представлялось
интересным изучить особенности
расположения комплексных сигналов в
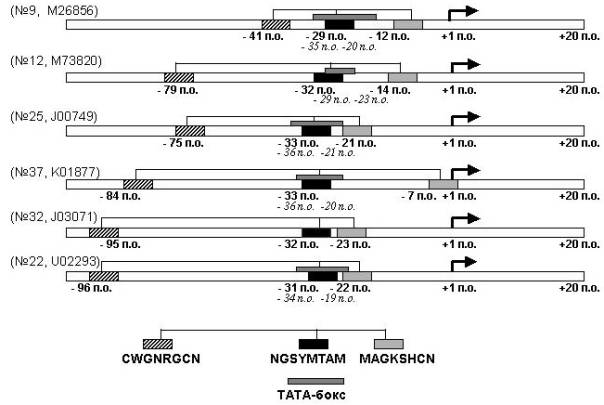
промоторах. На рисунке представлено
расположение описанного выше комплексного
сигнала CWGNRGCN<NGSYMTAM<MAGKSHCN в промоторах
генов эндокринной системы. Этот сигнал
выявлен в 6 промоторах и локализован в
районе от –100 п.о. до +20 п.о. относительно
старта транскрипции (последовательности в
выборке №9, 12, 22, 25, 32 и 37, идентификаторы EMBL:
M26856, M73820, U02293, J00749, J03071 и K01877. соответственно).
Этот
комплексный сигнал локализован в районе от
-95 п.о. до -7 п.о. относительно старта
транскрипции (отмечено положение первого
нуклеотида в олигонуклеотидном мотиве).
Положение TATA-бокса (Bucher, 1990), указанное в
базе данных TRRD, отмечено заштрихованными
прямоугольниками.
По рисунку легко заметить совпадение локализации второго олигонуклеотидного мотива с районом TATA-бокса. Из рисунка также видно, что в промоторах №№12,25 и 37 расстояние между первым и вторым и между вторым и третьим олигонуклеотидами комплексного сигнала составляет 42-51 и 12-26 п.о., соответственно.

Рис. Схема расположения комплексного сигнала CWGNRGCN<NGSYMTAM<MAGKSHCN в промоторах генов эндокринной системы (Витяев и др., 2001). Последовательности промоторов фазированы относительно старта транскрипции (позиция +1 п.о.), выделенного стрелкой. Номер исследуемого промотора указан слева в скобках. Входящие в комплексный сигнал олигонуклеотидные мотивы длиной 8 п.о. отмечены черными прямоугольниками, указана позиция первого нуклеотида относительно старта транскрипции. Положение TATA-бокса, проиндексированное в базе данных TRRD, отмечено заштрихованными прямоугольниками. Позиции первого и последнего нуклеотида в TATA-боксе указаны курсивом.
Также
близки между собой расстояния между этими
олигонуклеотидами в промоторах №22 и №32 (63-65
п.о. и 9 п.о.). В №9 эти расстояния составляют 12
и 17 п.о., соответственно. Первый
олигонуклеотид в комплексном сигнале –
CWGNRGCN – часто встречается в ССТФ Myogenin,
аннотированных в базе данных TRRD, третий
олигонуклеотид в сигнале – MAGKSHCN – в ССТФ Nrf2
(NF-E2-related factor 2), и средний по расположению
олигонуклеотид в сигнале соответствует TATA-боксу.
На
следующем рисунке приведен пример
расположения комплексного сигнала DNMYTTSA<DNYAADGG<RCAGMMDY в 8 последовательностях промоторов
эритроид-специфичных генов (номера в
выборке 8,9,10,11,14,16,17,25 и 39).
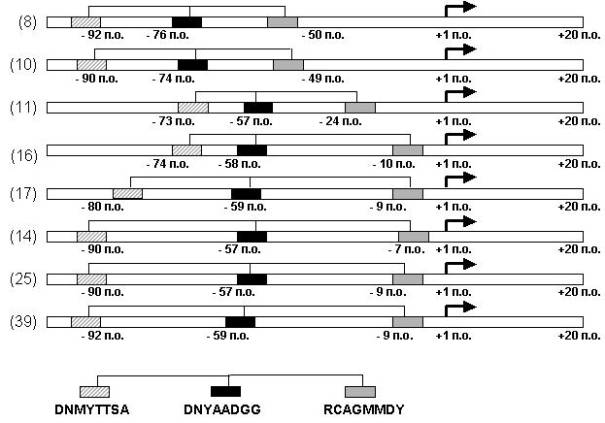
Рис. Пример расположения комплексного сигнала DNMYTTSA<DNYAADGG<RCAGMMDY в 8 последовательностях промоторов эритроид-специфичных генов (Витяев и др., 2001).
В этом
случае также выделяются характерные
расстояния между индивидуальными
сигналами. В промоторах №8,10 расстояние
между первым и вторым и вторым и третьим
олигонуклеотидами комплексного сигнала
равно 16 п.о. и 25-26 п.о., соответственно. В
промоторах №17, 14, 25 и 39 эти расстояния
составляют 21-33 п.о. и 50 п.о., соответственно (см.
рис. 3.3.2). Заметим, что первый олигонуклеотид
в сигнале DNMYTTSA встречается в сайтах связывания
транскрипционных факторов COUP и SF1,
аннотированных в базе данных TRRD. Эти
факторы участвуют в регуляции системы
эритропоэза.
В
дополнение к изучению промоторов эритроид-специфичных
генов и промоторов генов эндокринной
системы проведено исследование
нуклеотидных последовательностей
промоторов генов, экспрессирующихся в
других генных сетях: генов системы
липидного метаболизма, интерферон-регулируемых
генов и генов системы ответа на тепловой
шок. Последовательности указанных групп
промоторов, были извлечены из базы данных
TRRD (Kolchanov et al., 2002) и
фазированы
[-300;+100] относительно старта транскрипции.
Были
построены контрастные выборки
последовательностей, содержащих промоторы,
и последовательностей, не обладающих таким
свойством. По методике, описанной в
предыдущем разделе с помощью компьютерной
системы ARGO, разработанной О.В.Вишневским,
были построены наборы специфичных
олигонулеотидов в 15-буквенном алфавите для
каждой выборки последовательностей. Далее
с помощью системы Gene Discovery был выполнен
поиск групп олигонуклеотидов (комплексных
сигналов). Было найдено большое число
комплексных сигналов, содержащих пары и
тройки специфичных олигонуклеотидов.
Комплексные сигналы характеризуют, как
правило, 5-12 последовательностей (подгруппу
последовательностей из выборки).
Рассмотрим пример комплексного сигнала (YCTNNYTS,
DRVSCAG, WTAWWWR) для
промоторов генов системы липидного
метаболизма, найденного с помощью системы
Gene Discovery.
Regularity 203
IF
YCTNNYTS = 1 (Fisher criterion 0.004058)
AND
NDRVSCAG = 1 (Fisher criterion 0.005992)
AND
WTAWWWRN = 1 (Fisher criterion 0.020397)
THEN
Class = 1 (with frequency 9 / (0 + 9) = 1.000000)
Regularity
apply to objects: 2(+) 5(+) 16(+) 17(+) 24(+) 26(+) 31(+) 37(+) 50(+)
Рис.
Сигнал
{YCTNNYTS,
NDRVSCAG, WTAWWWRN}. Выдача программы.
Данный
комплексный сигнал (выдача программы)
включает три олигонуклеотида YCTNNYTS, DRVSCAG и WTAWWWR (символ N
– любой нуклеотид добавлен для
дополнения сигнала до стандартной длины 8
нуклеотидов). Графическое представление
дано на рисунке.

Рис. 3.3.4. Пример олигонуклетидных сигналов YCTNNYTS, NDRVSCAG, WTAWWWRN в 9 последовательностях промоторов генов системы липидного метаболизма. Олигонуклеотиды обозначены соответственно серыми, шрихованными и черными прямоугольниками. Идентификаторы генов в базе данных TRRD приведены слева, старт транскрипции выделен стрелкой.
Соответствие
идентификаторов последовательностей
имеющих показанный на рисунке
комплексный сигнал, функции и видовой
принадлежности последовательностей по
базе данных TRRD приведено ниже: A00158, A00349, A00293
– ген аполипротеин A-I трех организмов (Rattus
norvegicus, Oryctolagus cuniculus, Mus musculus), A00151 –
аполипротеин E человека,
A00888 – 25-гидроксивитамин D3-24-гидроксилаза
человека, A00409, A00376 и A00109
– ген CCAAT/enhancer binding protein alpha, (Rattus norvegicus, Homo
sapiens и Mus musculus, соответственно), A00264 –
аполипротеин AI человека.
Из
рисунка видно, что отдельные сигналы
встречаются по несколько раз в промоторной
последовательности, но наиболее
консервативным является расположение
относительно старта транскрипции -180, -110 и -30
п.о. соответственно для трех указанных
олигонуклеотидов. Наиболее консервативно
расположение сигнала WTAWWWR, удовлетворяющего
консенсусу TATA-бокса в районе -30 п.о.
относительно старта транскрипции (Bucher, 1990).
Олигонуклеотид
YCTNNYTS встречается в ССТФ GATA1, GATA2, Pu1, STAT1 (частота
встречаемости 0.3-0.4), а олигонуклеотид NDRVSCAG
встречается в ССТФ GATA3 и GATA4. Таким образом,
найденные
олигонуклеотидные сигналы в промоторах
могут соответствовать как эволюционно
консервативным участкам
последовательности, так и сайтам
связывания транскрипционных факторов.
Описание последовательности в 15-буквенном
коде не описывает полностью многообразие
последовательностей ДНК, связывающихся с
белками. Поэтому могут использоваться
другие методы предсказания сайтов,
учитывающие вес (представленность) каждого
нуклеотида в последовательности сайта
– в частности весовые матрицы,
составленные из последовательностей ДНК,
связывание которых с белками определено
экспериментально с помощью SELEX-экспериментов.
Рассмотрим
распределение специфичных
олигонуклеотидных сигналов для промоторов
других функциональных групп. На рисунке 3.3.5
показано расположение олигонуклеотидов в
промоторах генов ответа на тепловой шок.
Комплексный
сигнал определялся как совместное
присутствие олигонуклеотидов ARARAVNT и CYARAAVN.
Сигналы повторены по несколько раз, что в
целом характерно для промоторов генов
ответа на тепловой шок, имеющих по
несколько копий ССТФ. Из рисунка видно
упорядоченное расположение сигналов ARARAVNT и
CYARAAVN соответственно в районе -200 и -170
относительно старта транскрипции.
Олигонуклеотид CYARAAVN часто встречается в
сайтах связывания HSF1 –
фактора теплового шока (частота
встречаемости олигонуклеотида в
аннотированных сайтах TRRD 0.3).
В целом,
упорядоченное расположение характерно и
для других комплексных олигонуклеотидных
сигналов. И действительно, в аннотированных
промоторах расположение этого
олигонуклеотида совпадает с расположением
ССТФ HSF (Рис. 3.3.5, обозначено темным
прямоугольником под схематическим
изображением последовательности).

Рис. Пример олигонуклетидных сигналов ARARAVNT и CYARAAVN в 10 последовательностях промоторов генов ответа на тепловой шок, фазированных относительно старта транскрипции. Олигонуклеотиды ARARAVNT и CYARAAVN обозначены соответственно штрихованными и черными прямоугольниками. Идентификаторы EMBL генов приведены слева, старт транскрипции выделен стрелкой.
Рассмотрим
расположение комплексных
олигонуклеотидных сигналов в промоторах
интерферон-регулируемых генов. На рисунке
3.3.6 показано расположение комплексного
сигнала, включающего четыре вырожденных
специфичных олигонуклеотида NNMDVCTT, NHYYTNCC,
CWGRRWVN и SDNNKCWG.
Олигонуклеотид
NNMDVCTT соответствует ССТФ AP2, олигонуклеотид
NHYYTNCC часто встречается в
последовательностях ССТФ cKrox, Ets, NFkB и Pu1,
CWGRRWVN встречается в ССТФ EKLF, и, наконец,
олигонуклеотид SDNNKCWG встречается в ССТФ cKrox,
cMyb, ER, GATA2. Как показали исследования
последовательностей промоторов,
сгруппированных по функциональной
принадлежности и тканеспецифичности, все
они характеризуются наличием комплексных
сигналов, содержащих от двух до четырех
олигонуклеотидов.
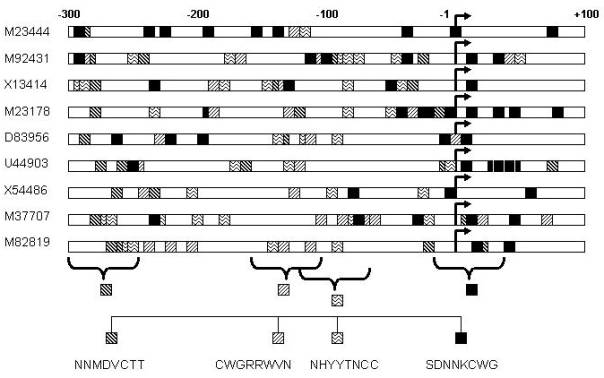
Рис. Пример комплексного сигнала совместного присутствия четырех олигонуклеотидов NNMDVCTT, NHYYTNCC, CWGRRWVN и SDNNKCWG в последовательностях промоторов интерферон-регулируемых генов. Олигонуклеотиды обозначены прямоугольниками с разной штриховкой и черного цвета. Обозначения на рисунке. Идентификаторы EMBL генов приведены слева, старт транскрипции выделен стрелкой. Показана предпочтительная локализация и общий паттерн относительного расположения олигонуклеотидов.
Такие
сигналы характерны для подгрупп
последовательностей. Как правило,
олигонуклеотиды повторены в нескольких
копиях, тем не менее, существует
предпочтительная локализация сигналов в
близких районах относительно старта
транскрипции.
Таким
образом, разработанная компьютерная
система "Gene Discovery" позволяет выявлять
как индивидуальные значимые мотивы (вырожденные
квазиинвариантные олигонуклеотиды), так и
комплексные сигналы. О функциональной
значимости комплексных сигналов
свидетельствует тот факт, что они имеют
сходное расположение в пределах подгрупп
специфичных промоторов (см. рисунки). Кроме
того, как отмечалось выше, комплексные
сигналы могут иметь сходные расстояния
между индивидуальными мотивами. При этом
анализируемые промоторы не имеют
выраженной гомологии.
Индивидуальные
мотивы могут соответствовать сайтам
связывания транскрипционных факторов. Было
показано, что они обогащены потенциальными
сайтами связывания транскрипционных
факторов по сравнению со случайными
последовательностями (Kondrakhin et
al., 1995). Индивидуальные мотивы могут также
соответствовать участкам ДНК,
обеспечивающим специфические
конформационные или физико-химические
свойства: повышенную гибкость ДНК,
легкоплавкость и т.д., необходимые для
функционирования промоторов.
При
рассмотрении комплексных сигналов следует
отметить несколько обстоятельств. Во-первых,
в ряде работ выявлены специфичные паттерны
распределения потенциальных сайтов
связывания транскрипционных факторов с
максимумами локализации различных сайтов в
различных участках промоторов. Таким
образом, наблюдающиеся комплексные сигналы
могут отражать преимущественное
расположение различных сайтов в
определенных участках промоторов (Zhang,
1998). Учет этого обстоятельства в работе (Kondrakhin
et al., 1995) позволил повысить точность
распознавания промоторов. В работах В.Г.Левицкого
(Левицкий и Катохин, 2001) выявлено разбиение
промотора на локальные участки с
характерным динуклеотидным составом.
Показано, что такие участки могут иметь
определенные конформационные или физико-химические
свойства. Таким образом, комплексные
сигналы могут иметь как контекстную, так и
смешанную контекстно-конформационную
природу, отражая присутствие в
определенных местах промотора, как
особенностей контекста, так и локальных
конформационных особенностей ДНК, значимых
для выполнения специфических функций
промоторов.
Во-вторых,
в последнее время активно изучается особый
тип регуляторных элементов, контролирующих
транскрипцию, которые называются
композиционными элементами (КЭ) (Kel et
al., 1995). Они образованы парами сайтов
связывания транскрипционных факторов (перекрывающимися,
сближенными либо удаленными друг от друга
на некоторое фиксированное расстояние),
которые в результате белок-белковых
взаимодействий между соответствующими
транскрипционными факторами приобретают
новые регуляторные свойства. Каждый из
сайтов в составе КЭ способен
функционировать по отдельности, но их
взаимодействие обеспечивает существенно
более выраженный активирующий или
репрессирующий эффект на транскрипцию гена.
Исследование закономерностей совместной
встречаемости и взаимного расположения
сайтов c помощью системы "Gene Discovery"
открывает путь для создания компьютерных
методов поиска потенциальных
композиционных элементов.
Общая методика распознавания на основе закономерностей описано в статье (Вишневский и Витяев, 2001). Каждой позиции анализируемой последовательности ставится в соответствие некоторый вес – функция, соответствующая предсказанию найти в локальном окружении этой точки искомый функциональный район. Такой суммарный вес соответствует вероятности найти такой же сигнал по случайным причинам. Подход расширен на комплексные сигналы. Для оценки вероятности получить комплексный сигнал в случайных последовательностях (из-за вырожденности олигонуклеотидов), можно использовать как компьютерный эксперимент, так и аналитические формулы. Вероятность P(S) наблюдать последовательность нуклеотидов S=X1X2...Xn оценивается как произведение вероятностей Xi.
P(S)=
![]() P(Xi) , i=1,2,...,n.
P(Xi) , i=1,2,...,n.
Выявление и учет комплексных сигналов в качестве дополнительной характеристики позволит повысить точность распознавания специфических групп промоторов в геноме. Результаты предсказания промоторов в геномной ДНК представлены в Интернете на сайтах авторов программ - исполнителей проекта.
Ю.Л.Орлов