Руководство
пользователя.
NatClass
Наименование операции
Построение классификации и анализа геномных
последовательностей.
Условия, при соблюдении которых возможно выполнение операции
Порядок выполнения операции
Подготовительные действия
Основные действия в требуемой последовательности.
1.
Загрузка
входных данных в программу.
Входными данными для программы являются две выборки последовательностей в формате FASTA: Positive Sequences (выборки геномных последовательностей, Negative Sequences (выборка случайных последовательностей, или контрастных геномных последовательностей).
Для загрузки обучающих данных
используется команда меню Source->Add Positive Sequences (Рис. 1) или кнопка ![]() панели инструментов. На экране появляется
мастер и предлагает указать имя файла с позитивной/негативной выборкой
последовательностей.
панели инструментов. На экране появляется
мастер и предлагает указать имя файла с позитивной/негативной выборкой
последовательностей.
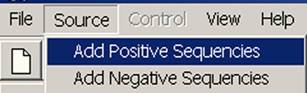
Рис. 1. Загрузка файла с позитивной выборкой последовательностей
В качестве входных данных могут служить файлы проектов, сохраненные ранее с помощью данной программы. В таких файлах проекта могут быть сохранены все данные, которые были загружены или получены на момент сохранения.
2. Установка параметров программы.
Запуск процесса генерации закономерностей.
На первой закладке, “Rules”, находятся элементы поиска закономерностей (рис.
2). Необходимо задать параметры поиска закономерностей и нажать кнопку “Пуск”.
Параметрами поиска являются:
Confidence interval: минимальный уровень
условной вероятности;
Min. Level of CP: порог для значения критерия Фишера;
Size of finish Buffer: количество
обнаруженных закономерностей;
Size of Sub Buffers: размер
вспомогательного буфера закономерностей.
Также выбирается режим работы: фиксированные
позиции (Fixed positions) или режим скользящего
окна (Shift positions). Последний
используется для распознавания вдоль длинной геномной
последовательности и требует указать размер окна (Width of scanning frame)
Программа позволяет приостановить процесс генерации закономерностей
(нажатием кнопки “Пауза”), или остановить процесс (нажатием кнопки “Стоп”).
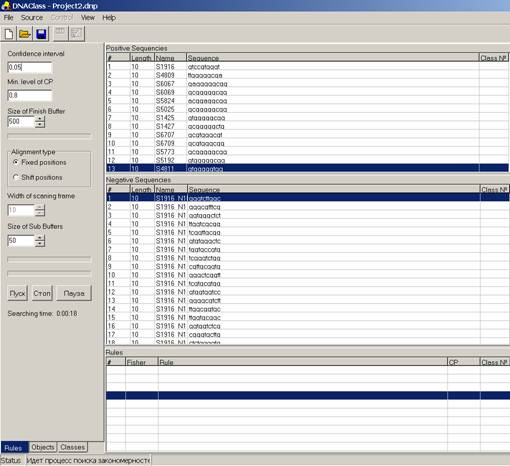
Рис. 2. Закладка элементов поиска закономерностей.
По окончании процесса
поиска закономерностей программа выдаёт сообщение “Процесс поиска
закономерностей успешно завершен”. В итоге найденные закономерности
представлены пользователю в порядке их обнаружения (Рис. 3).
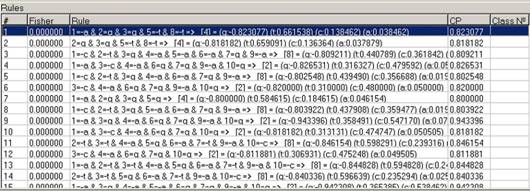
Рис. 3 Обнаруженные закономерности.
3.
Построение
идеальных объектов классов.
Кроме обнаруженных закономерностей, выходными данными программы NatClass так же являются идеальные представители классов. Для их построения служит закладка “Objects” программы (рис. 4).
Идеальные объекты могут строиться либо по начальным объектам из позитивной
обучающей выборки (опция “original objects”), либо по
закономерностям (“regularities”). Также можно выбрать
один из трех вариантов алгоритма построения (idealization type), задавая приоритеты между удалением и
добавлением признаков. После построения идеальных объектов, программа
соответственно относит объекты обучения к одному из обнаруженных классов, или
распознаёт их как принадлежащие к новому классу “New”. По аналогии с процессом генерации
закономерностей, процесс идеализации можно приостановить (нажатием кнопки “Пауза”), или остановить
(нажатием кнопки “Стоп”).
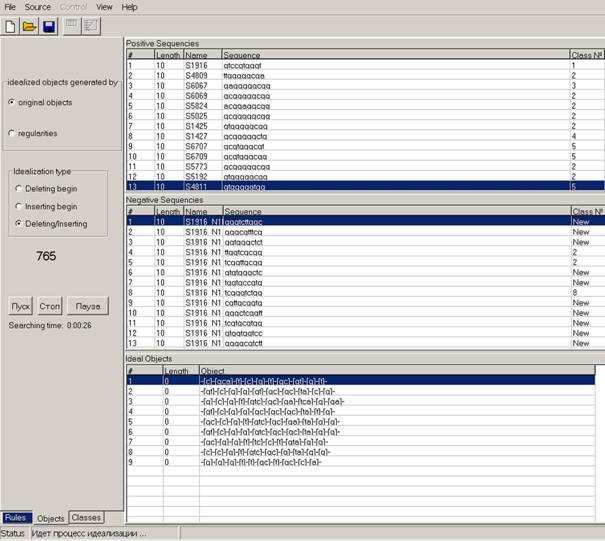
Рис. 4. Закладка элементов построения идеальных
объектов.
По окончании процесса поиска закономерностей программа выдаёт сообщение “Процесс идеализации успешно завершен”.
4.
Применение полученных закономерностей.
Вычисление ошибок распознавания.
Закладка Classes содержит функции
обработки полученных выходных данных (Рис. 5).
Здесь доступны следующие функции для анализа результатов счета: классификация контрольных выборок (“Classification”), распознавание относительно имеющихся классов (“Recognition Control Data”), подсчет ошибок распознавания (“Recognition Errors Count”), процедура Bootstrap.
Для загрузки контрольных
последовательностей используется команда меню Control->Add Control Positive.
При подсчете ошибок распознавания программа выдаст оптимальный
результат (построит гистограмму), но пользователь может самостоятельно
корректировать его, задавая либо порог распознавания (“Recogn Level”), либо значение ошибки первого рода (“1st
level error”).
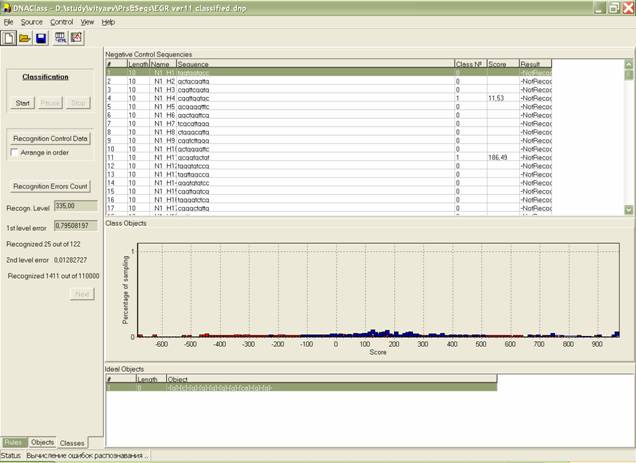
Рис. 5. Закладка “Classes”.
При нажатии правой кнопкой на идеальный объект появляется возможность
удалить объект (“Delete”, рис. 6), показать объекты класса (“Show Objects”, рис. 7), закономерности (“Show Regularities”), матрицу предсказания
(“Prediction matrix”),
матрицу распознавания (“Recognition Matrix”, рис. 8)
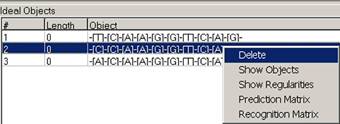
Рис. 6 Операции, производимые программой с
идеальным объектом.
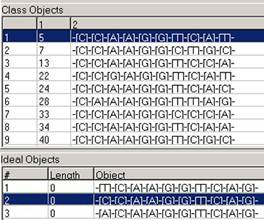
Рис. 7 Операция отображения объектов класса “Show objects”.
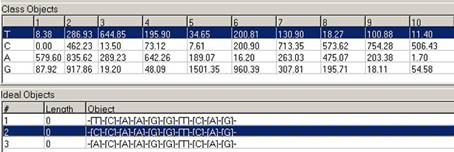
Рис. 8 Операция отображения матрицы распознавания
объектов класса “Recognition Matrix”.
Результаты распознавания
и ошибки сохраняются программой в виде html-таблицы.
Рекомендации по
освоению.
Контрольный пример: Построение
классификации и анализ сайтов связывания транскрипционного фактора (ССТФ) EGR1.
1.
Загрузка
входных данных в программу.
На вход программе в качестве позитивной выборки подаются ССТФ EGR1:
>S1916;
gtccgtgggt
>S4809;
ttgggggcga
>S6067;
gagggggcgg
…
файл
EGR1_pos.seq.
В качестве негативной – случайные последовательности,
сгенерированные с теми же нуклеотидными частотами, что и позитивные
последовательности:
>S1916; _N1_H1_W1;
gggtcttggc
>S1916; _N1_H2_W1;
gggcgtttcg
>S1916; _N1_H3_W1;
ggtgggctct
…
файл neg_2200.seq
Для загрузки входных данный см. Руководство пользователя,
пункт 1
2. Установка параметров
программы. Запуск процесса генерации закономерностей.
Установлены параметры поиска:
Confidence interval: 0,05;
Min. Level of CP: 0,8;
Size of finish Buffer: 2000;
Size of Sub Buffers: 100.
Программа обнаружила 2000
закономерностей (рис. 9).

Рис. 9 Закономерности, удовлетворяющие параметрам
поиска.
3. Построение идеальных объектов классов.
В результате работы
программы был обнаружен один класс. Идеальный объект класса и матрица
предсказаний приведена на Рис. 10.
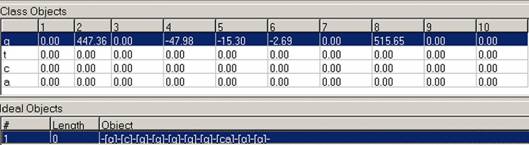
Рис. 10. Идеальный объект класса и матрица
предсказаний для ССТФ EGR1.
4.
Применение
полученных закономерностей. Вычисление ошибок распознавания.
В качестве негативного контроля были взяты последовательности,
сгенерированные с той же частотой нуклеотидов, что и позитивные
последовательности. Файл control_neg_1000.seq. Программой была проведена классификация,
вычисление веса каждого объекта, и распознавание (рис. 11).
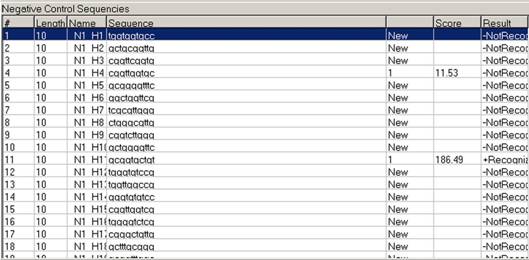
Рис. 11. Классификация и распознавание контрольных
объектов для ССТФ EGR1.